Respuesta de la bioinformática ante la pandemia de COVID-19
Romero Vivas, E. y Von Borstel Luna, F. D.
Dr. Eduardo Romero Vivas
Investigador Titular B
evivas@cibnor.mx
Centro de Investigaciones Biológicas del Noroeste, S.C.
Dr. Fernando Daniel Von Borstel Luna
Investigador Titular A
fborstel@cibnor.mx
Centro de Investigaciones Biológicas del Noroeste, S.C.
Introducción
El ambicioso Proyecto del Genoma Humano (PGH), inició el 1 de octubre de 1990, con la meta de secuenciar y mapear todos los genes que componen el genoma humano. En 2003, el PGH dio como resultado una secuencia preliminar terminada en un 99 por ciento, de más de 3 mil millones de pares de bases [1]. Esta compleja secuencia de pares de bases químicas compone el ADN (Ácido Desoxirribonucleico) de una persona promedio.
Por su parte, el virus SARS-CoV-2 responsable de la enfermedad COVID-19, reportado por primera vez en Wuhan, China, tiene una secuencia genómica de tan solo 29,903 pares de bases, y fue compartida el 5 de junio del 2020 a través de las bases de datos públicas de GenBank (MN908947.3/-NC_045512.2) por el gobierno de China, entonces denominado “Wuhan-Hu-1” [2]. Ésta secuencia fue anotada (es decir identificados sus componentes) basándose en la similitud con otros coronavirus.
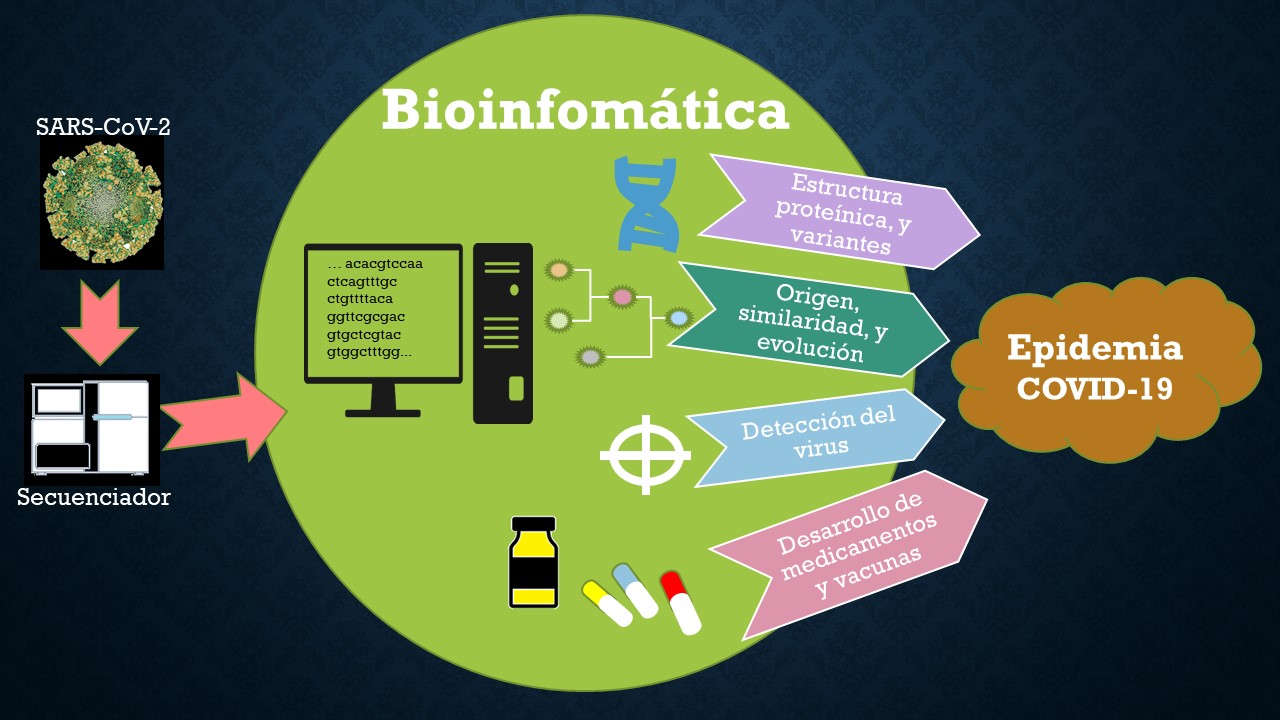
La secuenciación de ADN es el principal método en el diagnóstico del coronavirus y es una herramienta importante para la investigación del virus. Ésta consiste en determinar el orden de los componentes básicos (nucleótidos) en el código genético, y se logra mediante métodos y técnicas bioquímicas. Una vez obtenida esta secuencia ordenada de nucleótidos la información se codifica mediante cuatro letras A (adenina), C (citosina), G (guanina), T (timina), y se almacena como un texto. A partir de ahí se pueden realizar su análisis por medios informáticos, y se vuelve terreno de la bioinformática.
En el campo de la epidemiología y medicina en tiempos de COVID-19, la bioinformática, nos permite obtener un conocimiento profundo del genoma del virus, de las características de las proteínas que lo conforman, realizar diagnósticos clínicos para su detección, y desarrollar fármacos antivirales y vacunas.
El virus que causa el COVID-19 está cambiando constantemente y se espera que existan cada vez más variantes del virus. Estas variantes pueden desaparecer o permanecer, constituyéndose en nuevas cepas, que serán las bases para que surjan nuevas variantes conforme el virus se replica en los humanos y se propagan los contagios. Por ende, es importante monitorear las nuevas variantes y compararlas con las previas para determinar su grado de peligrosidad o que tan contagiosas son. Esta comparación se realiza a partir de sus propiedades, por ejemplo, la resistencia a un tratamiento, o a partir de su código genético obtenido por secuenciación y analizado mediante algoritmos computacionales a través de la bioinformática.
El análisis de esta información permite [3]:
- Conocer la estructura proteínica, las posibles variantes (al seguir los cambios de las secuencias), y su visualización.
- Conocer su origen, la similaridad con otros virus, y la posible evolución del mismo mediante la comparación de secuencias.
- La detección del virus, mediante herramientas de diagnóstico como el RT-PCR, su secuenciación en equipos de nueva generación, prueba de antígenos, entre otros. Lo cual es posible al conocer que secuencia de nucleótidos se debe buscar.
- El desarrollo de nuevos medicamentos mediante el entendimiento de la estructura tridimensional del virus, y evaluando las pequeñas moléculas de proteína que pueden combinarse con el virus para inactivarlo, es decir que estructuras lo forman y que estructuras le son afines usando aplicaciones de simulación y visualización.
- Utilizar diferentes aplicaciones para la investigación de vacunas y su efectividad.
Asimismo, una estrategia realizada por la comunidad científica para combatir la actual epidemia, fue implementar múltiples métodos de análisis en línea, para que la información pública relacionada a cómo afecta el virus a la población, pueda ser utilizada por usuarios que no sean expertos en computación. Algunas de estas aplicaciones de investigación y análisis, son creadas para navegadores de internet, con interfaces web interactivas [4]:
- Mapas epidemiológicos globales que recaban datos sobre el número de casos, muertes y recuperaciones, edad, hospitalizaciones, entre otros datos más, en una escala espacial y temporal.
- Aplicaciones enfocadas en compartir datos en tiempo real, en forma de hojas de cálculo interactivas, que colectan, adicionan y permiten análisis estadísticos sobre información demográfica de fuentes oficiales.
- Aplicaciones web interactivas que simulan la dinámica de la diseminación del COVID-19 y generan proyecciones en alguna población, basados en diferentes suposiciones epidemiológicas, así como los efectos de las medidas adoptadas para contener la transmisión de la enfermedad.
- Algunas aplicaciones web regionales para describir las curvas epidémicas y predecir los efectos de las políticas de reducción de interacción social sobre el número de casos y la capacidad de hospitales, colectar y visualizar datos epidemiológicos, entre otros análisis.
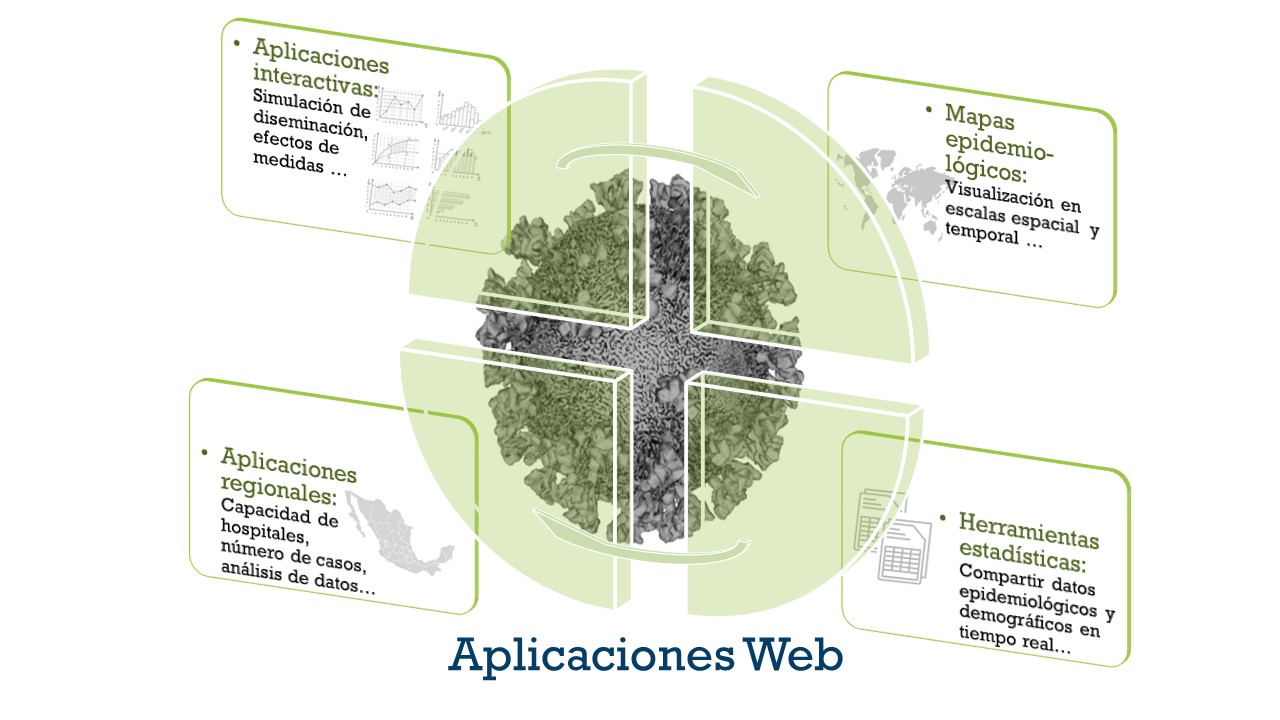
De esta manera, también se desarrollaron aplicaciones web de análisis genómico para los investigadores en todo el mundo, para realizar análisis de secuencias, y comparar el virus original con las muestras de casos locales, en búsqueda de mutaciones que permitan rastrear su origen de manera espacial y temporal, y tratar de explicar las diferencias en su transmisión en todos los países.
Conclusión
Para apoyar la investigación y el avance del conocimiento sobre el COVID-19 y su tratamiento, la comunidad bioinformática ha desarrollado aplicaciones en línea que permiten que una amplia audiencia pueda analizar los datos específicos del virus SARS-CoV-2, y la evolución de la pandemia a nivel global. De esta forma, han proporcionado un innegable apoyo a los actuales esfuerzos de investigación contra la pandemia, permitiendo un fácil acceso a información de suma importancia para la toma de decisiones clínicas, políticas y sociales.
Referencias:
[1] https://www.genome.gov/human-genome-project, visitado 20 de junio 2022.
[2] https://www.cdc.gov/museum/timeline/covid19.html, visitado 16 de junio 2022.
[3] L. Ma, H. Li, J. Lan, X. Hao, H. Liu, X. Wang, Y. Huang, Comprehensive analyses of bioinformatics applications in the fight against COVID-19 pandemic, Comp. Biol. and Chem., Vol 95, 2021, https://doi.org/10.1016/j.compbiolchem.2021.107599.
[4] D. Mercatelli, A. N. Holding, F. M. Giorgi, Web tools to fight pandemics: the COVID-19 experience, Brief. in Bioinformatics, Vol 22, Issue 2, March 2021, pp 690-700, https://doi.org/10.1093/bib/bbaa261
